Diseñan una IA para predecir la migración celular en el cáncer de mama
En este estudio, publicado en la revista Computers in Biology and Medicine, los investigadores han desarrollado una Inteligencia Artificial (IA) que supone un avance importante en la combinación de técnicas de deep learning y biología computacional.
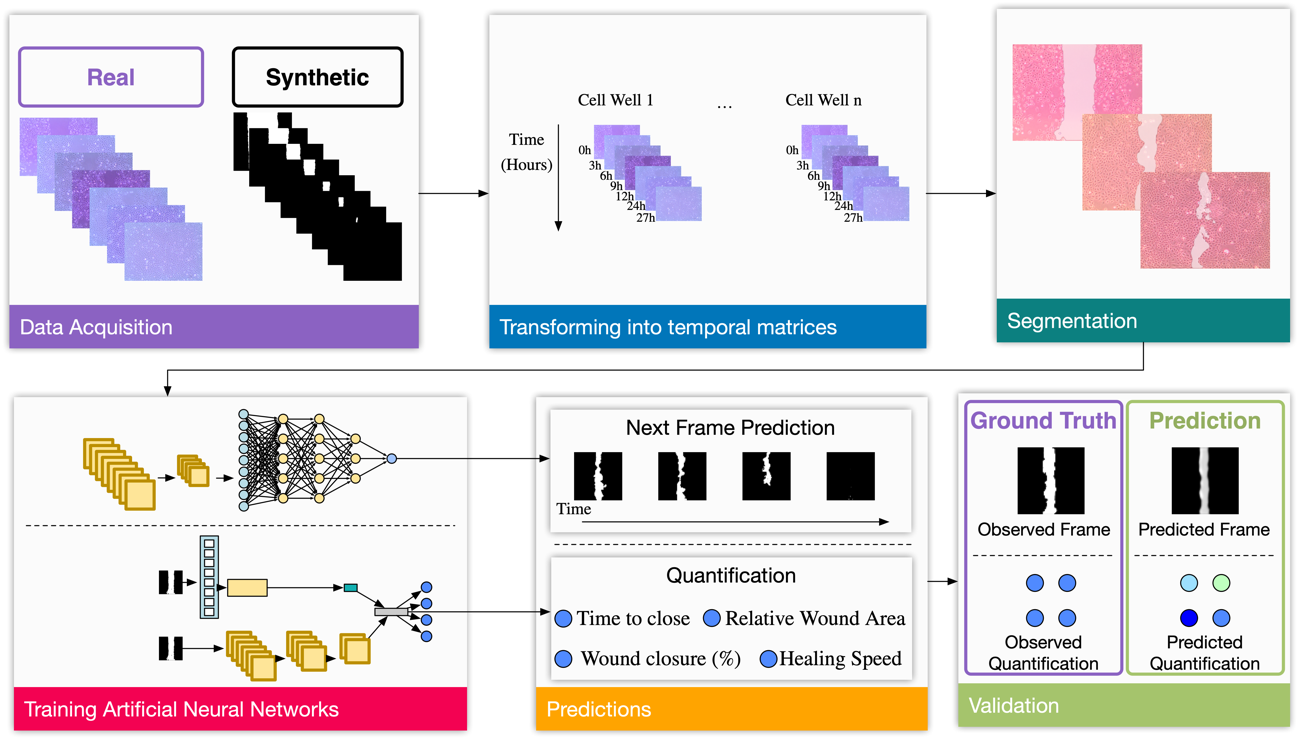
Un equipo de investigadores de la Universidad de Granada y la Universidad de Sevilla, liderados por Juan Antonio Marchal Corrales y Miguel Ángel Gutiérrez Naranjo respectivamente, ha publicado un estudio innovador en el que se diseña una Inteligencia Artificial para mejorar la predicción de la evolución de la migración celular en el cáncer de mama. El estudio, titulado Using Deep Learning for Predicting the Dynamic Evolution of Breast Cancer Migration, supone un avance importante en la combinación de técnicas de deep learning y biología computacional.
El trabajo multidisciplinar, con la participación de Francisco M. García Moreno y el doctorando Jesús Ruiz Espigares, ambos de la Universidad de Granada, se centra en el desarrollo de un marco predictivo denominado Prediction Wound Progression Framework (PWPF). Este marco aprovecha el poder del deep learning para analizar y predecir la migración celular en modelos bidimensionales —técnicamente conocidos por Wound Healing—, proporcionando nuevas perspectivas en la comprensión del proceso metastásico del cáncer de mama.
"La metástasis es la principal causa de mortalidad en pacientes con cáncer de mama y comprender cómo se produce la migración celular es crucial para desarrollar mejores estrategias terapéuticas", explica Jesús Ruiz, investigador co-principal del Departamento de Anatomía y Embriología Humana de la Universidad de Granada y miembro del Centro de Investigación Biomédica (CIBM).
El equipo ha desarrollado una arquitectura de redes neuronales basada en Conv-LSTM, que aprovecha tanto las características espaciales como temporales de los datos de migración celular. Esta arquitectura permite predecir con precisión la evolución de la técnica de Wound Healing a lo largo del tiempo, mejorando la capacidad para analizar la dinámica en el contexto de modelos de cáncer de mama. Este enfoque automatizado puede ser aplicado a modelos más complejos en 3D que mimeticen mejor las características de los tumores y promete abrir nuevas vías para la investigación y el tratamiento del cáncer.
La investigación es el resultado de una colaboración multidisciplinar entre diferentes departamentos y centros: el Departamento de Lenguajes y Sistemas Informáticos (LSI), el Departamento de Anatomía y Embriología Humana y el CITIC de la Universidad de Granada, el Laboratorio Singular BioFabi3D_Biofabricación y (bio)impresión 3D del CIBM, la Unidad de Excelencia «Modeling Nature» y el Instituto de Investigación Biosanitaria ibs.GRANADA, además del Departamento de Ciencias de la Computación y la Inteligencia Artificial de la Universidad de Sevilla.
El avance del equipo no sólo destaca por su contribución científica, sino también por su accesibilidad y promoción al acceso abierto, ya que el código y los datos generados están disponibles públicamente en sus repositorios de GitHub y Zenodo, fomentando el acceso abierto y la colaboración internacional en la investigación del cáncer.
El proyecto ha sido llevado a cabo gracias a la financiación del Ministerio de Ciencia, Innovación Universidades (MICIN), la Consejería de Salud Familias de la Junta de Andalucía y la Cátedra Doctores Galera y Requena de Investigación en Células Madre Cancerígenas de la UGR
Referencia bibliográfica:
Garcia-Moreno FM, Ruiz-Espigares J, Gutiérrez-Naranjo MA, Marchal JA. Using deep learning for predicting the dynamic evolution of breast cancer migration. Comput Biol Med. 2024 Sep;180:108890. doi: 10.1016/j.compbiomed.2024.108890. Epub 2024 Jul 27. PMID: 39068903.
